original high-fidelity enzyme
PCRBIO HiFi Polymerase PCRBIO HiFi Polymerase は、3’-5’ エキソヌクレアーゼ (校正) 活性を持つ Pfu DNA ポリメラーゼから派生したPCR Biosystems 社オリジナルの高忠実度酵素です。
いくつかの独自の突然変異点により、ネイティブ型と比較してパフォーマンスが大幅に向上します。高度なバッファー化学と組み合わせることで、この酵素は高忠実度 PCR の世界に強力なパフォーマンスをもたらします。
Taq DNA ポリメラーゼ より長いアンプリコンによる PCR 成功率の向上
Mg や dNTP を含む高度なバッファー化学
標準および高速 PCR 条件下で高収量
GC リッチおよび AT リッチ配列を含む複雑なテンプレートからの効率的かつ特異的な増幅
高忠実度 PCR
ブラントエンドクローニング
部位特異的突然変異誘発
コロニーPCR
「難しい」PCR : GC/AT リッチ DNA
PCRBIO HiFi Polymerase とは? PCRBIO HiFi Polymerase は、DNA ポリメラーゼ技術とバッファー化学の最新開発を使用して、PCR の速度、収量、特異性を高めます。この酵素とバッファー システムは、哺乳類のゲノム DNA などの複雑なテンプレートで優れた PCR パフォーマンスを発揮します。DNA 結合が強化されたため、処理能力が向上し、収量が増加し、サイクル時間が短縮されます (5kb 増幅産物の 35 サイクルを 1.5 時間未満で実行)。PCRBIO HiFi Polymerase は、GC と AT リッチの両方を含む幅広いテンプレートで一貫して良好なパフォーマンスを発揮します。
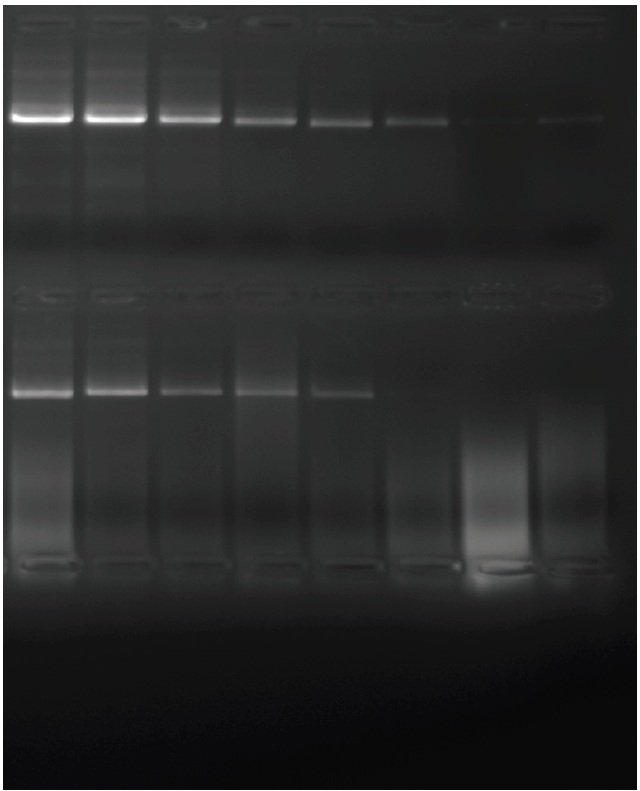
参考データ1:精製されたヒトゲノム DNA に由来する GAPDH からの 5 kb アンプリコンの増幅
精製されたヒトゲノム DNA に由来する GAPDH からの 5 kb アンプリコンの増幅を示します。2倍のテンプレート希釈系列を、開始濃度100ナノグラムのDNAから作製しました。30 秒の変性、30 秒のアニーリング、75 秒の伸長の 25 サイクルが 1 時間で完了しました。最初の行は PCRBIO HiFi Polymerase 、2 行目は Finnzymes の同等製品、3 行目は標準 Pfu を示しています。
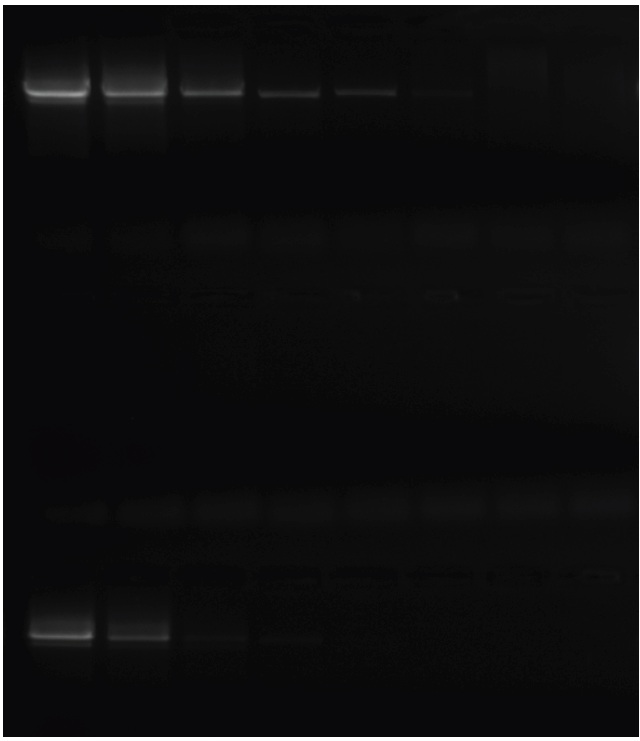
参考データ2:ゲノム DNA からヒト GAPDH の 60% GC 1kb フラグメントを増幅
PCRBIO HiFi Polymerase がゲノム DNA からヒト GAPDH の 60% GC 1kb フラグメントを増幅する様子を示しています。テンプレートは 100 ナノグラムから始まり、8 桁にわたって 2 倍に希釈されます。最初の行は PCRBIO HiFi Polymerase 、2 行目は Invitrogens Pfx 酵素、3 行目は Finnzymes Phusion 酵素を示しています。
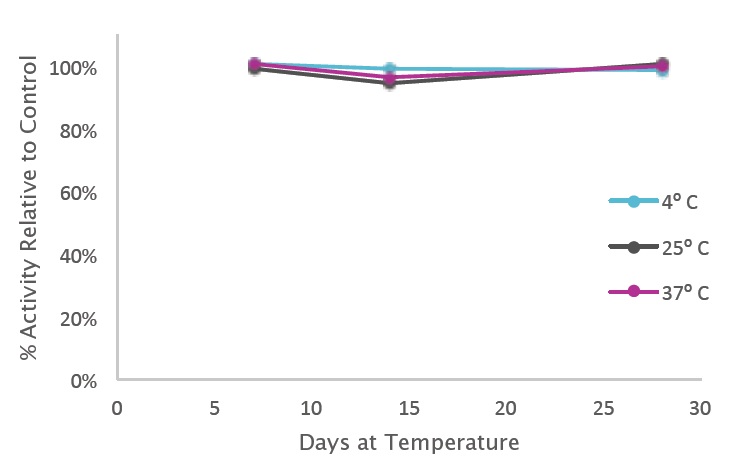
4℃、25℃、37℃で 28 日間放置した後でも、PCRBIO HiFi ポリメラーゼ で活性の変化が検出されないことを示します。
PCRBIO HiFi Polymerase
アッセイに特殊なバッファーが必要な場合、PCRBIO HiFi Polymerase を使用できますか?
PCRBIO HiFi Polymerase に付属する 5x PCRBIO HiFi Buffer は、この酵素専用に開発されたものであり、併用することを強くお勧めします。ただし、PCRBIO HiFi Polymerase は、野生型 Taq で使用するために開発されたあらゆる PCR バッファーと互換性がある必要があります。PCRBIO HiFi Polymerase でカスタマイズしたバッファーを使用する場合は、アニーリング温度や酵素、テンプレート、dNTP、MgCl2 の濃度などの反応パラメータの最適化が必要になる可能性があることに留意してください。
PCRBIO HiFi Polymerase はコロニー PCR に使用できますか?
はい。細菌コロニーを使用する場合は、滅菌チップを使用してコロニーを選択し、50 µL の PCR 反応液に再懸濁します。液体培養から作業している場合は、最終混合物に 5 µL の一晩培養液を追加します。一般的なプロトコルに従い、最初の変性時間を 95 °C で 10 分に増やします。
PCRBIO HiFi Polymerase はマルチプレックス PCR に使用できますか?
PCRBIO HiFi ポリメラーゼ にはホット スタート テクノロジが含まれていませんが、低温では本質的に活性が低く、ホット スタートのない Taq ポリメラーゼと比較してはるかに低いです。つまり、一部のマルチプレックス アプリケーションに使用できます。
マルチプレックス PCR を初めて実行する場合、55°C から 65°C のアニーリング温度勾配を実行することをお勧めします。最高の特異性が得られるアニーリング温度を、その後の実験で使用する必要があります。マルチプレックス PCR には高速サイクリング条件を使用しないでください。最初は 90 秒の延長時間を推奨しますが、収量を増やすためにこの時間を延長することもできます。
マルチプレックス反応の場合、反応は最初から最後まで氷上または冷却ブロック上で行う必要があります。プライマーは、末端検出法で簡単に分析できる多様なアンプリコン長を維持しながら、重複する配列をできるだけ避けるように慎重に設計する必要があります[1]-[3]。PCRBIO HiFi Polymerase のアンプリコンの長さの制限に注意してください。アンプリコンの 1 つが限界に近い場合は、VeriFi® Polymerase
Markoulatos, P., Siafakas, N. & Moncany, M. Multiplex polymerase chain reaction: a practical approach. J Clin Lab Anal 16 , 47-51, doi:10.1002/jcla.2058 (2002).
Radhika, M., Saugata, M., Murali, H. S. & Batra, H. V. A novel multiplex PCR for the simultaneous detection of Salmonella enterica and Shigella species. Brazilian Journal of Microbiology 45 , 667-676, doi:10.1590/s1517-83822014005000041 (2014).
Perez-Perez, F. J. & Hanson, N. D. Detection of plasmid-mediated AmpC beta-lactamase genes in clinical isolates by using multiplex PCR. J Clin Microbiol 40 , 2153-2162, doi:10.1128/jcm.40.6.2153-2162.2002 (2002).
PCRBIO HiFi Polymerase は校正できますか?
はい。PCRBIO HiFi Polymerase は 3’-5’ エキソヌクレアーゼ (校正) 活性を持っています。
私の結果には、非特異的なアンプリコンやスメアのバックグラウンドが多く含まれています。トラブルシューティングについて何かアドバイスはありますか?
スメアが懸念される場合は、それが最適ではない条件でアガロースゲル電気泳動を実行したことによるアーティファクトではないことを確認することをお勧めします。最適ではない条件には、高電圧やゲルが固まるのに十分な時間を与えないことが含まれます[1]。
また、PCR 反応のトラブルシューティングを行い、以下の提案を検討する必要がある場合もあります [2]。
プライマーは、プライマー間の相互作用を防ぎ、特異性を向上させるように設計する必要があります
アニーリング温度を上げるか、アニーリング温度勾配 PCR を実施して最適なアニーリング温度を決定します
反応中のテンプレートの量を減らします。高品質の DNA を得るには、50 µL 反応あたり 1 ~ 100 ng のゲノム DNA または 5 ng 以下のプラスミド / ラムダ DNA を使用します
サイクル数を減らします
反応あたりの酵素量を減らします
プライマー濃度を下げますが、各プライマーの濃度は 100 nM 以上にしてください
最終濃度が 5% ~ 10% になるように反応液に DMSO を含めます
Koontz, L. Agarose Gel Electrophoresis. Laboratory methods in enzymology : DNA . First edition. edn, Vol. 529 35-45 (2013).
Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp , e3998, doi:10.3791/3998 (2012).
私の結果では、収率が非常に低いことがわかりました。トラブルシューティングについて何かアドバイスはありますか?
以下の提案を検討し、文献[1]も参照するとよいでしょう。
アニーリング温度勾配 PCR におけるアニーリング温度を最適化します
反応中のテンプレートの量を増やしてください
サイクル数を増やしてください
反応あたりの酵素の量を増やします
プライマー濃度を増やしますが、各プライマーの濃度が 1 μM を超えないようにしてください
新鮮なdNTP溶液を試してください
MgCl2 を最適化します
Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J Vis Exp , e3998, doi:10.3791/3998 (2012).
PCRBIO HiFi Polymerase と VeriFi® Polymerase の違いは何ですか?
VeriFi® Polymerase PCRBIO HiFi Polymerase に比べて処理能力が強化されており、伸長時間が短く (10 ~ 30 秒/kb)、収量が高く、より長い (最大 17.5 kb) より困難なターゲットを増幅できます。また、VeriFi® Polymerase PCRBIO HiFi Polymerase よりも忠実度が高くなっています。PCRBIO HiFi Polymerase とは異なり、VeriFi® Polymerase
PCRBIO HiFi Polymerase のエラー率はどれくらいですか?
この酵素のエラー率は、取り込まれたヌクレオチド 4.5 x 107 あたり約 1 個のエラーです。これは、PCRBIO Taq DNA polymerase
PCRBIO HiFi Polymerase の推奨伸長時間はどれくらいですか?
真核生物 DNA からの増幅には 1 キロベース (kb) あたり 30 秒が推奨されます。ウイルスや細菌の DNA では伸長時間を短縮できます。
PCRBIO HiFi Polymerase を使用して生成されたフラグメントには、どのようなタイプのクローニング戦略を使用すればよいですか?
PCRBIO HiFi Polymerase によって生成された PCR 産物は平滑末端を持つため、それ以上の改変を必要とせずに平滑末端クローニングに適しています。
A-オーバーハングを追加する場合は、Taq DNA polymerase または Klenow (exo-) DNAを使用し、その後TAクローニングを行います[1]。A-オーバーハングの追加を開始する前に、PCRBIO HiFi Polymerase が反応液から除去されていることを確認してください。PCRBIO HiFi Polymerase が存在する場合、酵素の3’-5’エキソヌクレアーゼ活性によりAオーバーハングが除去されます。
Yao, S., Hart, D. J. & An, Y. Recent advances in universal TA cloning methods for use in function studies. Protein Eng Des Sel , doi:10.1093/protein/gzw047 (2016).